令和2年6月26日掲載
 PCRって何?
PCRって何?
ポリメラーゼ連鎖反応(polymerase chain reaction PCR)は、DNA(デオキシリボ核酸(Deoxyribonucleic acid)は、核酸の一種)を増幅するための原理またはそれを用いた手法で、手法を指す場合はPCR法と呼ばれることの方が多いが、ポリメラーゼチェーンリアクションとも呼ばれる。
次の特徴を持つ。
ヒトのゲノム(約30億塩基対)のような非常に長大なDNA分子の中から、自分の望んだ特定のDNA断片(数百から数千塩基対)だけを選択的に増幅させることができる。しかも極めて微量なDNA溶液で目的を達成できる。増幅に要する時間が2時間程度と短い。
PCR法そのものや派生した様々な技術は、分子遺伝学の研究のみならず、生理学・分類学などの研究にも広く応用されている。また、医療や犯罪捜査にも大きな役割を果たしている。特に近年では感染症分野での病原体診断に大きく貢献している。
 1.感染症分野での応用
1.感染症分野での応用
感染症分野特にウイルス感染症の診断に関しては、ウイルスがDNAだけあるいはRNAだけしか持たないため、RNAウイルスに関してはRNAの検出が必要でありそのためにはRNAをDNAに変換する必要がある。そこで開発されたのが逆転写ポリメラーゼ連鎖反応(reverse-transcriptase PCR)である。
 2.原理
2.原理
|
2本鎖DNAは、水溶液中で高温になると、変性し1本鎖DNAに分かれる(図1①Denaturation)。変性が起こる温度は、DNAの塩基構成および長さ(塩基数)によって異なり、長いDNAほど高い温度が必要になる。 急速に冷却すると、長いDNA同士は2本鎖に再結合しにくいが、短いDNA断片(オリゴヌクレオチド)は結合できる。 プライマーがDNAよりも圧倒的に多い状況にしておくことで、DNA-プライマーの結合がDNA-DNAの結合より、さらに優先的になる。 以上述べてきたようにPCR法は、DNA鎖長の違いによる変性とアニーリングの違いを利用して、温度の上下を繰り返すだけでDNA合成を繰り返し、DNAを増幅する技術である。 PCR法開発当初はDNA変性の時にDNAポリメラーゼが失活するためサイクル毎に酵素を追加していたが、現在ではTaqポリメラーゼ(好熱菌Thermus aquaticus)など好熱菌の耐熱性DNAポリメラーゼ(EC.2.7.7.7)を用いることで連続して反応を進めることができる。 |
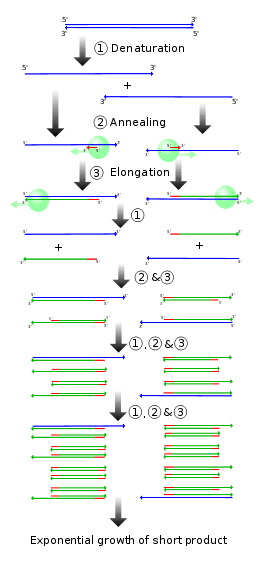 図1 PCR法の原理 |
 3.留意点
3.留意点
この反応の成否は、増幅対象DNAとプライマーの塩基配列、サイクル中の各設定温度・時間などに依存する。それらが不適切な場合、無関係なDNA配列を増幅したり、増幅が見られないことがある。
また、合成過程において変異が起こる可能性も少なからずあるため、使用目的によっては生成物の塩基配列のチェックが欠かせない。
 4.PCR法の処理フロー
4.PCR法の処理フロー
(1) ターゲットとするウイルスがDNAウイルスであるかRNAウイルスであるかによって、反応液調整までの操作が異なる。
(1)-1 DNAの抽出
① 細胞を破壊 (溶解) する。その方法には以下の3パターンがある。
- SDSなどのionic detergentsを使う方法。
- グアニジン塩など、分子の疎水結合を弱め水溶性を高めるionic detergentsを使う方法。
- アルカリとionic detergentを組み合わせる方法。
② DNA とタンパク質の結合を切断。
- プロテアーゼを使用する方法。
- DNAを吸着するマトリックスを使う方法。
- フェノール/フェノクロ処理などでタンパク質を変性させて取り除く方法。
③ 最後にエタノール沈殿で塩を除く必要がある。
(1)-2 RNAの抽出と逆転写反応
細胞や組織からRNAを抽出する方法は現在、様々なキットが出回っている。AGPC(acid guanidinium thiocyanate-phenol-chloroform extraction)法の原理を示す。DNAやRNAは酸性の環境において、そのリン酸基の電離平衡がO-からOHへと偏る。リン酸基の電荷が打ち消された結果、DNAは電気的に中性になり、無極性溶媒に溶けやすくなる。しかし、RNAは、そのリボースにOH基があるため、DNAに比べて親水のような極性溶媒に溶けやすくなっているため、組織や細胞の破砕液に酸を加えて酸性にした後、フェノール、クロロホルムなどの無極性溶媒を加えると、DNAやタンパク質は無極性な有機層へ移行するのに対して、RNAは水層に留まる。この水層を単離して、アルコール沈殿を行うことでRNAを精製できる。精製したRNAは逆転写ポリメラーゼ連鎖反応(Reverse Transcription Polymerase Chain Reaction, RT-PCR)といってRNA を鋳型に逆転写を行い、生成された cDNA に対して PCR を行う方法で、RNAを鋳型としてDNA を合成する働きを持つ酵素(逆転写酵素)を用いてRNAをcDNAに変換する。
(2) 反応液調整
増幅対象DNA、プライマー※、DNAポリメラーゼおよびDNA合成の素材(基質)であるデオキシヌクレオチド三リン酸(dNTP)、そして酵素が働く至適塩濃度環境をつくるためのバッファー溶液を混合し、PCR装置にセットする。
※増幅対象のDNA領域の両端の塩基配列を決定し、対応するプライマーを人為合成する。 このときプライマーは、増幅予定の2本鎖DNAの両鎖それぞれの3'側に結合する相補配列であり、通常20塩基程度。
(3) PCRサイクル
| ア | 反応液を94℃程度に加熱し、30秒から1分間温度を保ち、2本鎖DNAを1本鎖に分かれさせる。 |
| イ | 60℃程度(プライマーによって若干異なる)にまで急速冷却し、その1本鎖DNAとプライマーをアニーリングさせる。 |
| ウ | プライマーの分離がおきずDNAポリメラーゼの活性に至適な温度帯まで、再び加熱する。実験目的により、その温度は60-72℃程度に設定される。DNAが合成されるのに必要な時間、通常1分から2分、この温度を保つ。 |
| ここまでが1つのサイクルで、以後、アからウまでの手順を繰り返していく事で特定のDNA断片を増幅させる。 | |
一般的にPCR処理をn回のサイクルを行うと、1つの2本鎖DNAから目的部分を2n倍に増幅すると言われている。 しかし、図1を見て分かる通り、特定部分だけが抽出されるまでには少なくとも3サイクル必要であり、長いDNA鎖も最後まで残ってしまうため、これは厳密に言えば正確ではない。
ただ、通常は20サイクル程度行う事から、これらの量は無視される。
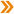 5.リアルタイムPCR法とは
5.リアルタイムPCR法とは
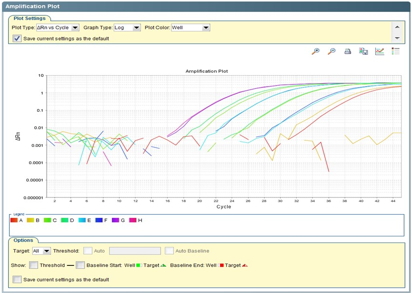
リアルタイムPCR法とは、PCRの増幅量(増幅曲線)をリアルタイムでモニターし解析する方法であり、迅速性と定量性に優れている。
 |
 |
|
図3 リアルタイムPCRシステム |
図4 増幅曲線 |
(1) インターカレーター法
二本鎖DNAに結合することで蛍光を発する試薬(インターカレーター:TB Green,SYBR Greenなど)をPCR反応系に加える方法である。インターカレーターは、PCR反応によって合成された二本鎖DNAに結合し、励起光の照射により蛍光を発します。この蛍光強度を検出することにより、増幅産物の生成量をモニターできます。また、増幅DNAの融解温度を測定することもできます。
(2) TaqMan法
PCRにより増幅したDNAに蛍光標識したプローブが結合すると蛍光が発し、その蛍光強度を検出する方法でもっとも広く使われているのがTaqMan法である。TaqManプローブは遺伝子特異的な配列に設計され、PCRプライマー間のターゲットに結合するようにデザインされている。TaqManプローブの5′末端にはターゲットの増幅をレポートする蛍光色素である、「レポーター」が結合し、プローブの3′末端にはレポーターからの蛍光を消光するクエンチャーが結合している。クエンチャーは、プローブの3′末端をブロックして熱安定性DNAポリメラーゼによる伸長を阻止する役目も果たしている。
 6.歴史と背景
6.歴史と背景
シータス社のキャリー・マリスが車でガールフレンドと夜道をドライブ中に、当時すでに知られていたオリゴヌクレオチドとDNAポリメラーゼを用いたDNA合成反応を繰り返すことにより核酸の一定領域を増幅することを思いつく。

マリスはこの方法を"polymerase-catalyzed chain reaction"(ポリメラーゼ触媒連鎖反応)と名付け、ネイチャー、サイエンスなどの著名な科学雑誌に論文として投稿したが、掲載されなかった。1987年にようやく、その論文は Methods in Enzymology 誌に掲載された。
一方、PCR法自体はシータス社の同僚の手により鎌状赤血球症という遺伝性疾患の迅速な診断手段に応用された。サイエンス誌に"Enzymatic
amplification of beta-globin genomic sequences and restriction site analysis
for diagnosis of sickle cell anemia"として報告され(1)、オリジナル論文より前に世界の科学者の注目を集めることとなった。
PCRは、当初、E.coli のDNAポリメラーゼIをズブチリシン処理し5'-3'エキソヌクレアーゼ活性を除去したクレノー断片を用いて反応を起こすものが大半であった。この酵素を用いた場合は、二本鎖DNA変性のための温度上昇の際に、DNAポリメラーゼが失活し、サーマルサイクルごとに手作業でこの酵素を加えなければならなかった。
シータス社の研究グループは、この欠点を解決するために好熱性の細菌から得た耐熱性DNAポリメラーゼを用いたPCRを開発し、サイエンス誌に発表した(2)。これにより、PCR反応の簡便化、自動化への道が開かれ、幅広く応用可能な手法として発展することになった。
このように、PCR法の応用、発展にはシータス社グループ(当初はマリスも含む)のはたした役割が大きいのであるが、最初にこの方法を着想し、方向性を示したという業績により、1993年にキャリー・マリスがノーベル化学賞を受賞している。
 7.参考文献
7.参考文献
(1) Saiki, R. K.; Scharf, S.; Faloona, F.; Mullis, K. B.; Horn, G. T.; Erlich, H. A.; Arnheim, N. (1985). "Enzymatic amplification of beta-globin genomic sequences and restriction site analysis for diagnosis of sickle cell anemia." Science 230 (4732): 1350-1354. PMID 2999980.
(2) Saiki, R. K.; Gelfand, D. H.; Stoffel, S.; Scharf, S. J.; Higuchi,
R.; Horn, G. T.; Mullis, K. B.; Erlich, H. A. (1988). "Primer-directed
enzymatic amplification of DNA with a thermostable DNA polymerase."
Science 239 (4839): 487-491. PMID 2448875
神奈川県衛生研究所第19代所長
髙崎 智彦

